|
|
|
|
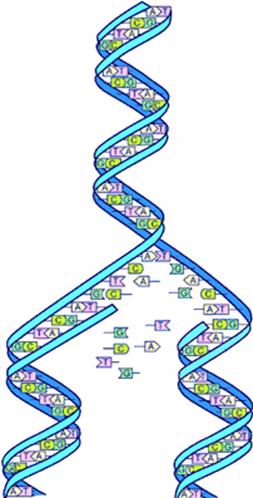
Replikacja DNA to proces, w którym podwójna nić DNA ulega skopiowaniu. Replikacja jest semikonserwatywna - w każdej z dwóch uzyskanych podwójnych nici DNA będzie jedna nić macierzysta i jedna nowa. Nie licząc niewielkiego prawdopodobieństwa (ok. 1 błąd na 109 nukleotydów, dla porównania błąd transkrypcjii - 1 na 104) wystąpienia błędu obie cząsteczki DNA będą identyczne. Substratami tego procesu są: matryca DNA; trifosforany deoksyrybonukleotydów (dNTP); ATP - energia dla helikaz; W procesie tym stwierdzono wiele aktywności enzymatycznych (udział enzymów) tj.: helikazy - rozrywają wiązania wodorowe między nićmi matrycowego DNA, rozkręcając helisę i umożliwiając rozpoczęcie procesu; prymaza - syntetyzuje starter; polimerazy DNA - polimeryzuje zgodnie z zasadą komplementarności fosforany deoksyrybonukleotydów; egzonukleaza - usuwa startery RNA z nici; ligaza DNA - uzupełnia brakujące wiązania fosfodiestrowe w szkielecie nowo-zsyntezowanej nici DNA różne enzymy pomocnicze Zasady replikacji są podobne u wszystkich organizmów, przy czym największe różnice występują między bakteriami z jednej strony, a archea i eukariontami z drugiej. U bakterii replikacja zaczyna się w ustalonym miejscu i postępuje bardzo szybko, z prędkością rzędu 1000 nukleotydów na sekundę. U eukariotów replikacja jest o wiele wolniejsza, ok. 50 nukleotydów na sekundę, jednak zachodzi równocześnie w wielu miejscach. Polimeraza DNA działa jedynie w kierunku od końca 3' do końca 5' (czyli syntetyzuje nową nić w kierunku od 5' do 3'). Z tego powodu jedna z nici jest syntezowana w sposób ciągły, druga (ta, którą chcielibyśmy zsyntezować w przeciwną stronę) fragmentami (tzw. fragmenty Okazaki). |
Szczegóły procesu Kopiowanie podwójnej helisy DNA jest procesem złożonym. Proces dzieli sie na fazy inicjalizacji, wydłużania i terminacji. W kolistych cżąsteczkach DNA replikacja rozpoczyna się w miejscu inicjacji, liczącym ok. 200-300 par nukleotydów. Miejsce to oznacza się skrótem ori od ang. origin. W linernych chromosomach aktywnych jest wiele(tysiące) procesów jednoczesnej replikacji. Aby replikacja przebiegła prawidłowo, rozdzielenie obu nici musi zajść bez zniszczenia ich struktury podstawowej (I-rzędowej) i w odpowiednich warunkach, jak: dokładne odczytanie matrycy DNA, obecność odpowiedniej liczby wolnych nukleotydów, zachowania komplementarności. Na koniec musi dojść do terminacji replikacji, ewentualnego uzupełnienia braków na końcu nowopowstałej cząsteczki i połączenia nowych cząsteczek w helisę. U bakterii zakończenie replikacji jest niemal automatyczne (po skopiowaniu całego kolistego DNA, który jest pojedynczym replikonem. U eukariotów miejsc replikacji (replikonów) jest wiele, a jej terminacja zachodzi po zakończeniu wielu procesów replikacyjnych zachodzących niemal jednocześnie w różnych miejscach replikujących cząsteczek DNA. Do terminacji dochodzi, gdy widełki replikacyjne replikonu natkną się na specjalną sekwencję terminacyjną. Proces replikacji nie-kolistych (eukariotycznych) cząsteczek DNA wiąże się z problemem wolnych zakończeń powstających cząsteczek DNA. Zakończenia te, zwane telomerami składają sie z krótkich, ale wielokrotnie powtórzonych sekwencji. Replikazy wydłużają jedynie istniejące już nici, nie są natomiast w stanie zsyntetyzować końcowych odcinków telomerów. W rezultacie odcinki te narażone są na regularne skracanie. Skracaniu temu zapobiega obecność telomerazy, która przeprowadza odwrotną transkrypcję tych odcinków, posługując się jako "matrycą" nie DNA, ale RNA, będącym częścią składową tego enzymu. Zapobiega to odsłonięciu znaczących fragmentów DNA. Grupa białek rozwijających widełki replikacyjne to prymosom.
|